聚类算法——Clustering by fast search and find of density peaks
这是14年发表在Science上的一篇文章,论文中提出了一种基于密度的聚类算法,这种算法不需要指定类别的个数。聚类中心基于数据的局域密度峰给出,并且聚类中心一旦确定后就不会再迭代。
整个算法是下面这个流程。
1. 寻找密度峰
对于每一个数据点,计算它的局域密度\(\rho_i\): \[ \rho_i = \sum_j \chi(d_{ij}-d_c) \]
其中\(\chi\)是Heaviside阶梯函数,\(d_c\)是一个选定的阈值。从式子可以看出\(\rho_i\)等于距离点\(i\)距离小于等于\(d_c\)的点的个数。也可以使用指数衰减计算: \[ \rho_i = \sum_j e^{-\left(\frac{d_{ij}}{d_c}\right)^2} \]
2. 计算\(\delta_i\)
\(\delta_i\)定义为局域密度比\(\rho_i\)高的点到点\(i\)的最小距离。也就是从如果从\(i\)出发,那么至少走\(\delta_i\)的距离才能找到第一个密度比\(i\)高的点: \[ \delta_i = \mathop{\mathrm{min}}\limits_{j:\rho_j>\rho_i} d_{ij} \] 对于密度最大的点,令\(\delta_i\)等于它到最远的点的距离$_i=j d{ij} \(例如,数据如图A所示。计算\)_i\(和\)_i$,并分别以它们为横轴和纵轴,绘于图B。
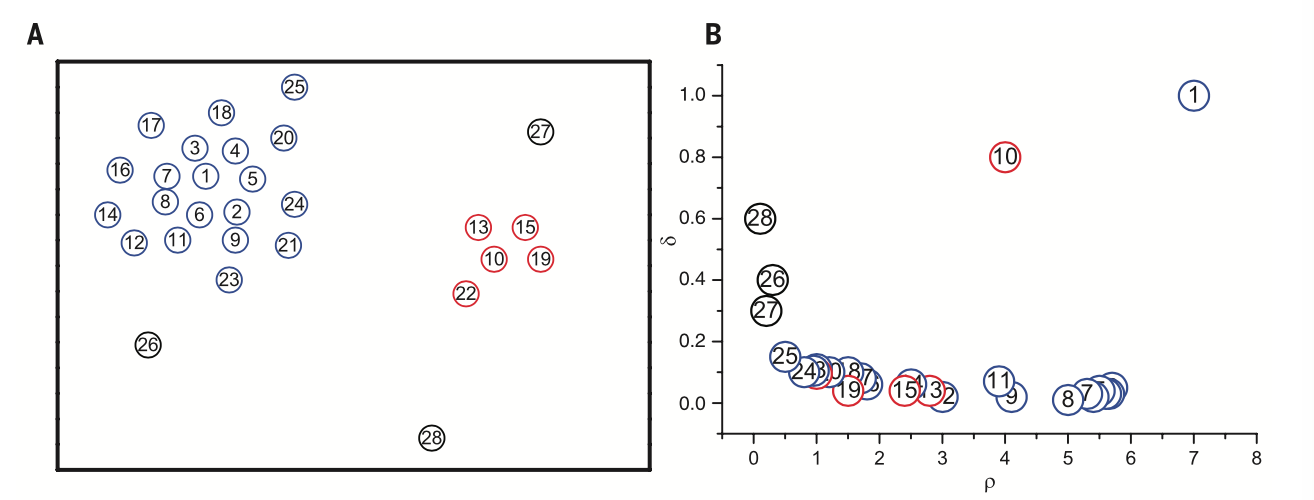
clusterAB
3. 选择聚类中心
聚类中心的特点是\(\rho_i\)和\(\delta_i\)都比较高,既有高密度,又离“潜在对手”比较远。在图B中为1和10两个点。可以看出聚类中心在决策图(图B)上明显地与其他点分离。通过这种方式,可以较轻易地把聚类中心挑出来。图B还包含了其他信息。例如,左上方的点是密度低又离其他点远的,这些点一般是噪声点。
4.聚类
在确定了聚类中心后, 其它点依据局部密度从高到低排列,依次确定所属的类别。每个点的类别为距离最近的且密度高于该点的点所属的类别,这是一个逐步扩张的过程。这样的方式使得数据的聚出的类可以反映数据的真实分布,这是相对于kmeans的一大优势,因为如果数据分布非球形,kmeans是很难聚出非球形类的。
参数选择
这个算法对\(d_c\)的选择较不敏感。作者建议升序排列所有的点点距离,然后取前1%到2%分位数作为\(d_c\)。或者使得平均邻居数是总点数的1%到2%。
选择聚类中心时还要指定两个阈值\(\rho_c\)和\(\delta_c\),可以参数化为 \[ \begin{align*} \rho_c &= \rho_{min}+\alpha (\rho_{max}-\rho_{min})\\ \delta_c &=\delta_{min} + \beta(\delta_{max}-\delta_{min}) \end{align*} \] 根据上图看,\(\alpha\)和\(\beta\)取0.15左右似乎就可以有不错的效果。或者画出决策图,人工挑选参数。
各个数据上的实验的图就不贴了,可以参见原文。
实现代码
|
|
参考文献
[1] Clustering by fast search and find of density peaks, Science, Alex Rodriguez and Alessandro Laio.